近期,西北工业大学生命学院施建宇教授与计算机学院于会副教授组成“BT+IT”交叉学科研究团队在药物-药物相互作用(DDI)预测方法与分析取得了进展。该研究团队在生物信息类国际顶级期刊BriefingsinBioinformatics(中科院1区Top期刊,中国自动化学会推荐的T1级生物信息期刊,IF=11.6)上发表了题为“STNN-DDI:a Substructure-aware Tensor Neural Network to predict Drug-Drug Interactions”的研究性论文。该研究成果得到了国家自然科学基金和和陕西省重点研发计划等科研项目的资助。文章在线链接为:https://doi.org/10.1093/bib/bbac209。下文简要介绍了其主要内容。
当多个药物同时被服用,药物之间可能会发生意料之外的相互作用(Drug-Drug Interaction, DDI)。DDI对治疗造成的影响既可能有害,也可能有益。有害DDI会降低药物的疗效、引起严重副作用、甚至增加意想不到的毒性,会使得那些使用由多种不同药物组成的药物复方进行治疗的患者处于危险治疗当中。另一方面,有益DDI却会产生“1+1>2”的治疗增效作用,这种DDI也被称为增效药物组合,可以成为一种安全有效的复杂疾病治疗方法,已被成功地应用到癌症和艾滋病等复杂疾病的治疗中。然而,由于代谢动力层级和药物动力层级的复杂机制,导致DDI的类型高达上百种,并可能引发多种不利作用。因此,分析DDI所引发的生物效应模式并提供相应的预测工具,就显得至关重要。这不仅能够筛选有害DDI,同时也能推断增效药物组合,从而应用于复杂疾病治疗。
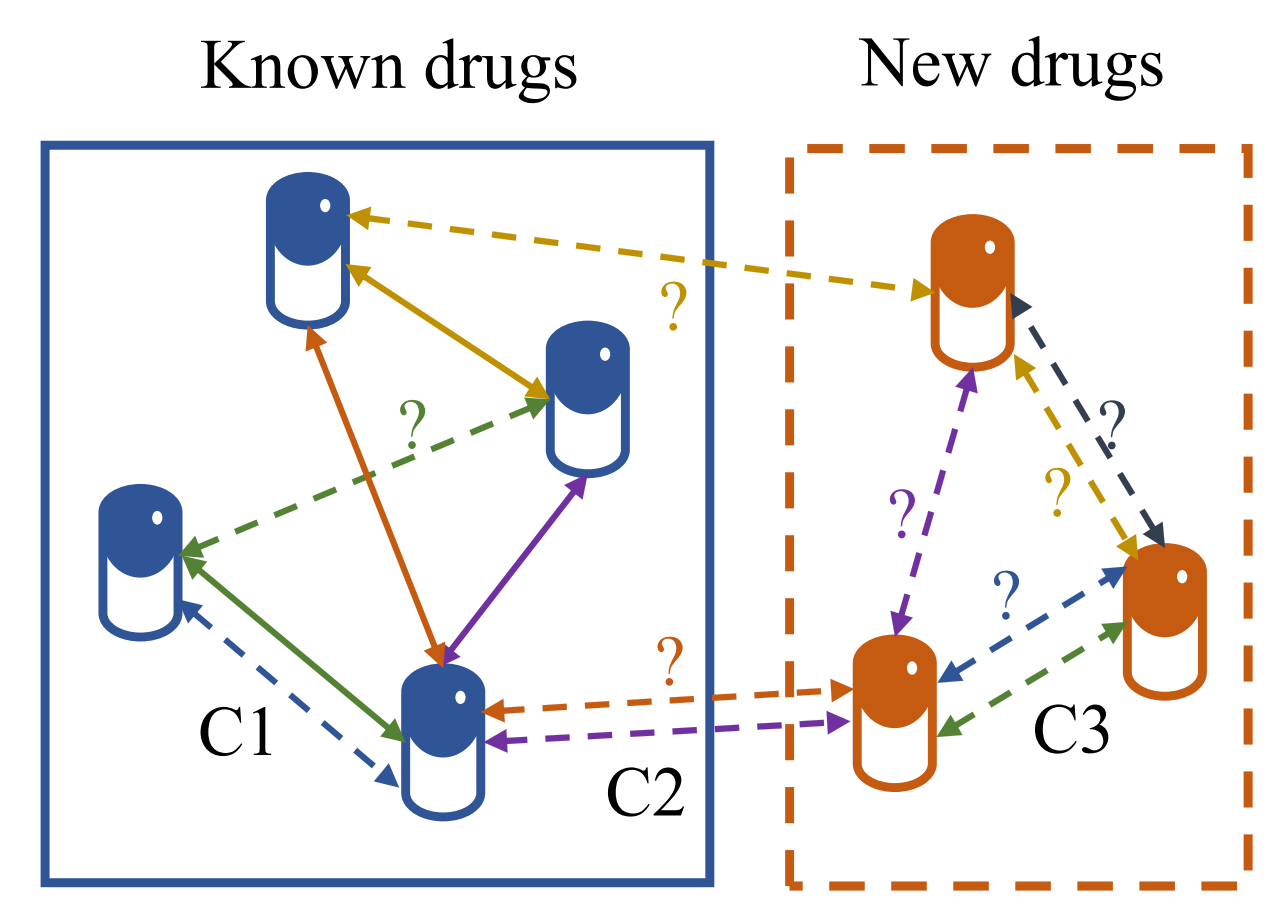
图1复杂类型“药物-药物”相互作用
尽管现有基于机器学习的DDI预测方法已经取得了一定进展,但是仍然存在两个主要问题:(a)仅预测两种药物之间产生的特定类型相互作用,无法判别两类药物之间可能产生的多种类型相互作用;(b)难以揭示引发DDI的关键药物局部结构,不利于揭示DDI产生机制。
本研究团队假设DDI是由药物的局部化学结构(子结构)引起的,其相互作用类型是由不同的子结构成对组合所决定的,进而设计了一个基于子结构感知的张量神经网络模型来分析和预测DDI(简称STNN-DDI,如图2所示)。该模型从化学子结构角度阐述了药物相互作用中的两个问题:(a)引发某种特定DDI类型的成对关键子结构(图3);(b)两个药物之间发生多重相互作用时,每种类型各自对应的关键子结构对(图4)。此外,提供了一个高精度的多类型DDI预测模型。总之,STNN-DDI为预测DDI以及解释药物相互作用的产生机制提供了一种有效方法。(文/图:施建宇;审核 杨慧)
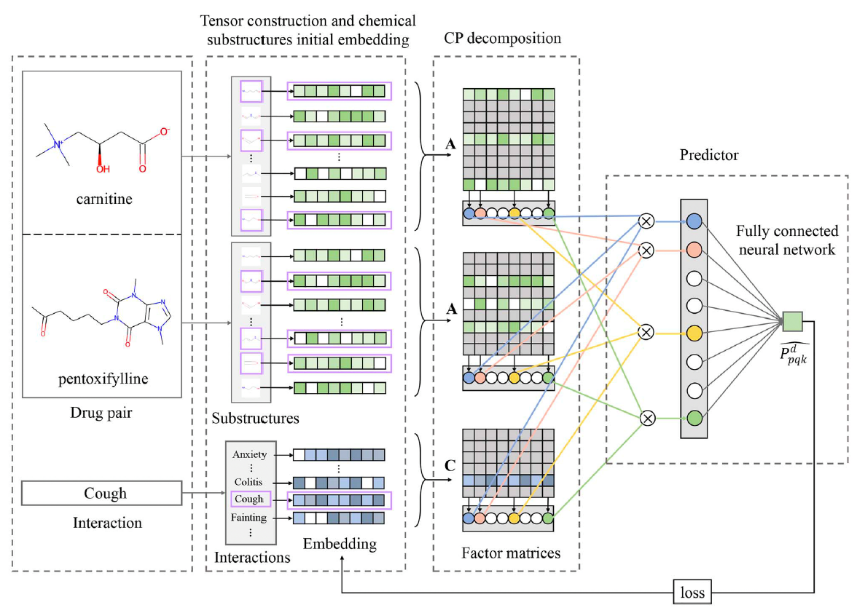
图2. STNN-DDI模型流程图
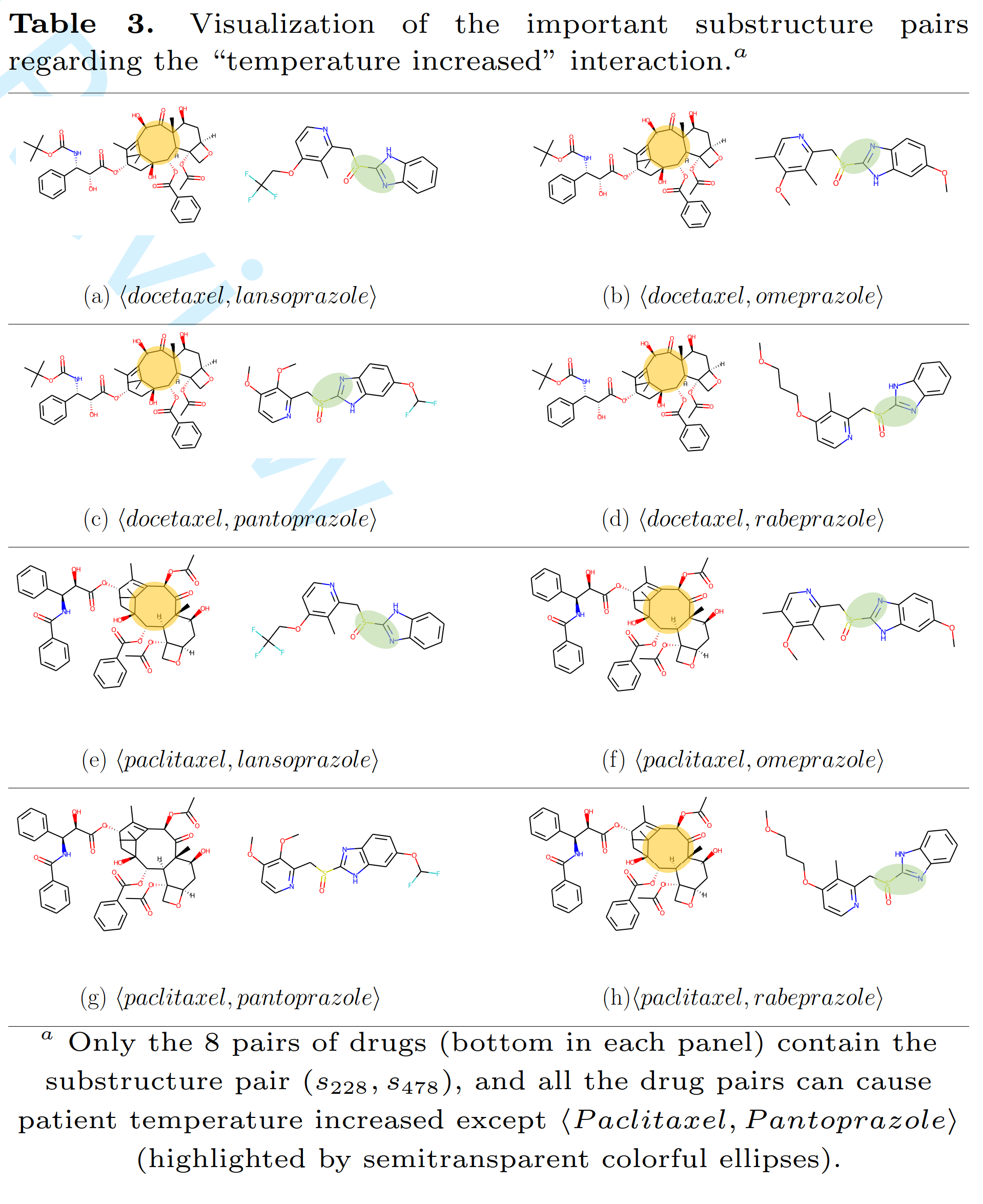
图3.特定DDI类型的成对关键子结构
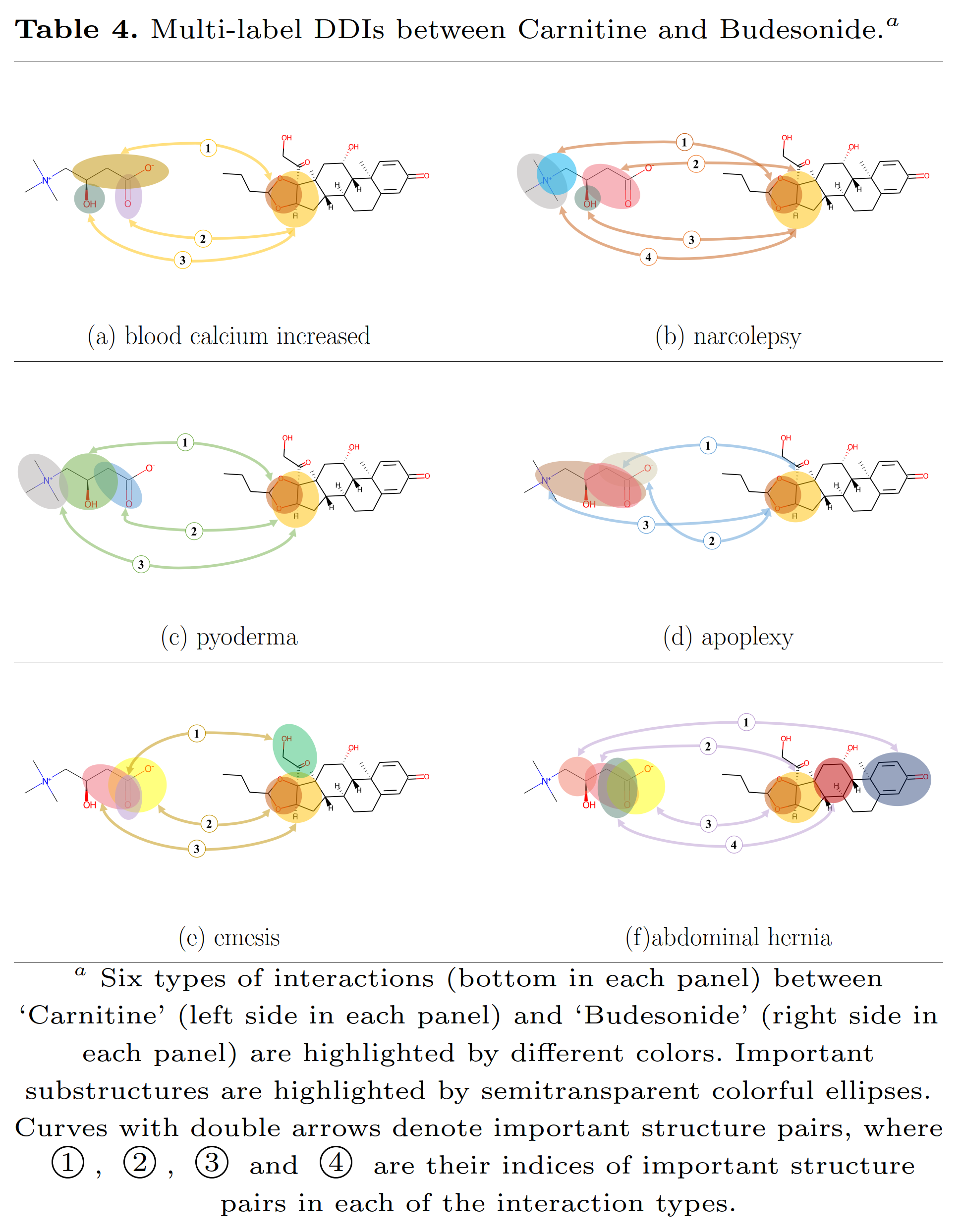
图4.两个药物之间发生多重相互作用时,每种类型各自对应的关键子结构对