近期,西北工业大学生命学院施建宇教授与新加坡信息通讯研究所Min Wu 资深科学家合作在生物信息学期刊《Bioinformatics》上发表了题为 “CMMS-GCL: cross-modality metabolic stability prediction with graph contrastive learning” 的研究性论文。西北工业大学生命学院为该论文的第一完成单位和通讯单位,博士生杜冰雪为该论文的第一作者,施建宇教授和Min Wu 资深科学家为该论文的共同通讯作者。该研究成果得到了国家自然科学基金、陕西省重点研发计划以及CAAI华为MindSpore基金等科研项目的资助。
下文简要介绍了本文的研究内容。
1. 研究背景
在新药研发的初期阶段,代谢稳定性的评估对于药物候选物的筛选和先导化合物的优化发挥着至关重要的作用。药物的代谢稳定性影响其生物利用度、半衰期和毒性等,因此准确预测分子的代谢稳定性对于降低开发成本和提高研发效率具有重大潜力。然而,传统的方法进行代谢稳定性的评估既耗时且昂贵,这限制了药物发现过程中高效筛选候选药物的能力。
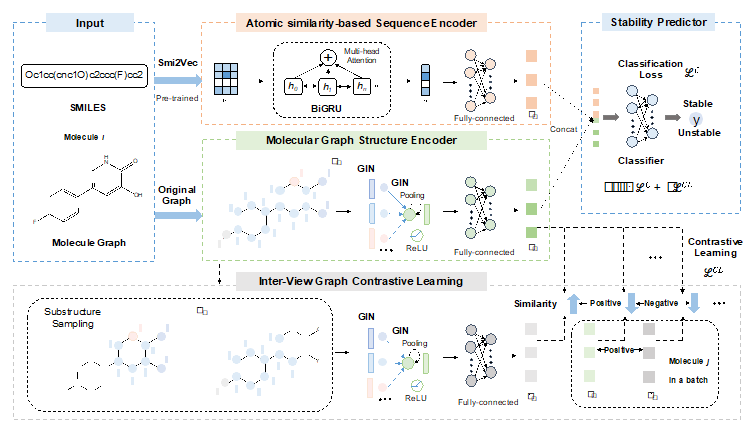
图1 CMMS-GCL模型框架
2. 研究方法
为了解决这些问题,本研究提出了CMMS-GCL模型,这是一个跨模态图对比学习方法,用于预测药物候选物的代谢稳定性。该模型结合了SMILES序列数据和分子图数据的深度学习技术,提出了一种新颖的方法来从两种不同的数据形式中提取特征,并通过图对比学习来进一步增强分子图特征。CMMS-GCL包含基于原子相似性的序列编码器、分子图结构编码器、图对比学习以及稳定性预测器(图1)。CMMS-GCL模型在多个数据集上的测试结果表明,它在预测准确性和解释性方面均优于其他先进方法,为药物发现和候选物的筛选提供了新的工具。
3. 可解释性分析
统计性分析部分对CMMS-GCL模型进行了深入的可解释性评估。本研究通过分析模型识别的关键官能团,并将这些官能团与已知的药物代谢数据库进行比对,来验证模型的预测准确性。分析结果表明,CMMS-GCL不仅能够准确预测代谢稳定性,而且能够清晰地指出哪些化学结构特征对代谢稳定性起着决定性作用。此外,模型还能够揭示出分子的哪些特定部分与药物代谢过程中的关键酶相互作用,从而进一步加深了对药物代谢机制的理解。这项统计性分析为CMMS-GCL模型的解释能力提供了强有力的证据,并为未来的药物设计和优化提供了有价值的指导。

图2 CMMS-GCL关键官能团对代谢稳定性影响的统计性分析
4. 参考文献
Bing-Xue Du, Yahui Long, Xiaoli Li, Min Wu*, Jian-Yu Shi*. CMMS-GCL: Cross-Modality Metabolic Stability Prediction with Graph Contrastive Learning. Bioinformatics. 2023,39(8): btad503 (Q1, 2023 IF=5.8,中国自动化学会T1级期刊)。
文章链接:https://academic.oup.com/bioinformatics/article/39/8/btad503/7241687。
(文/图:施建宇、杜冰雪;审核:杨慧)